Crear medicamentos nuevos es un camino difícil y, no voy a mentirles, también muy caro. Además, la mayoría de las veces, no se obtienen los resultados esperados. Pero no todas son malas noticias; han surgido técnicas de cribado virtual capaces de mejorar la eficiencia y reducir el costo de este largo y difícil proceso. Sin embargo, todavía estamos aprendiendo a usarlas de la mejor manera para aumentar su precisión y eficiencia.
Existen diferentes tipos de estas técnicas de cribado virtual. Algunas se basan en la estructura de las proteínas. Un ejemplo de estas es el acoplamiento molecular, donde un programa por ordenador intenta predecir la posición de unión óptima y también calcula una puntuación de acoplamiento para que los diferentes compuestos acoplados puedan clasificarse entre sí. Otro tipo de cribado virtual es el basado en ligandos, donde el objetivo es encontrar compuestos con propiedades químicas similares a las de un ligando objetivo. Ejemplos de esto son la detección farmacofórica y la búsqueda de similitud de huellas dactilares. El último método principal de cribado virtual se basa en algo de lo que todos hemos oído hablar alguna vez: la inteligencia artificial. Las técnicas basadas en estructuras y ligandos ya llevan tiempo en el ruedo y se han aplicado en muchos contextos. Sin embargo, el cribado utilizando la Inteligencia Artificial es el “recién llegado” y ha experimentado un gran aumento en popularidad y eficacia en los últimos años. El principio básico es bastante sencillo: utilizamos una gran cantidad de datos de laboratorio (por ejemplo, datos de actividad IC50) y los empleamos para entrenar modelos de aprendizaje automático. Una vez entrenados los modelos, podemos utilizarlos para examinar bibliotecas virtuales y comprobar qué compuestos se prevé que sean activos. Lamentablemente, las cosas no suelen ser tan sencillas, y normalmente hay muchos más pasos y complejidad en juego.
Por ejemplo, ¿cómo podemos representar una molécula y sus características de forma que un ordenador pueda entenderla y trabajar con ella? Una forma de hacerlo es convirtiéndolas en lo que llamamos huellas dactilares o descriptores moleculares. Pensad en las huellas dactilares moleculares como el DNI de la estructura de una molécula. Existen muchos métodos diferentes para codificar la estructura de una molécula en una huella dactilar. Un ejemplo son las huellas dactilares de subestructura, como las claves MACCS o la huella dactilar Pubchem. En la imagen 1 puedes ver un ejemplo de cómo se componen estas huellas dactilares.
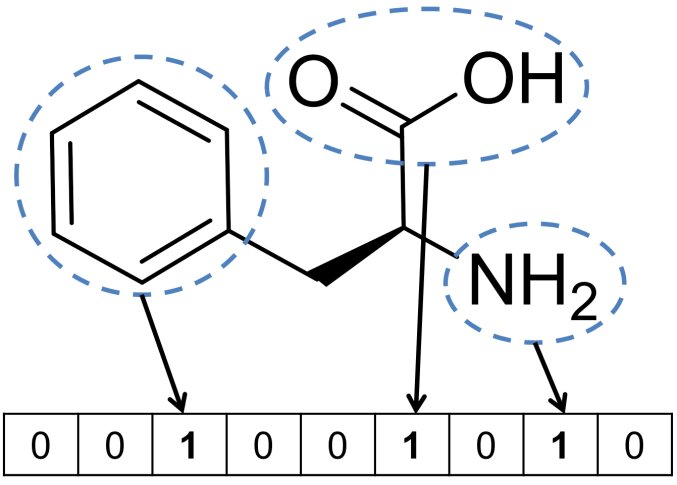
Ejemplo de una huella dactilar de subestructura. Hay características predefinidas que representan diferentes subestructuras. Si una determinada subestructura está presente, el valor de esa característica se establece en 1; de lo contrario, permanece en 0. (Figura tomada de https://doi.org/10.1016/B978-0-12-409547-2.12345-5)
Otro tipo son las huellas hash, como las huellas de conectividad extendida (ECFP). Existen muchas más huellas dactilares, pero todas tienen como objetivo representar la estructura de una molécula y sus características. Esto permite compararlas entre sí (búsqueda de similitudes), pero también las hace utilizables para entrenar modelos de aprendizaje automático.
Hoy en día existen muchas arquitecturas diferentes de aprendizaje automático, desde las más sencillas, como los modelos de regresión lineal y los árboles de decisión, hasta modelos más avanzados, como las redes neuronales artificiales. Muchos de estos modelos avanzados pueden dar lugar a predicciones muy precisas. Pero aquí viene el problema: a menudo se consideran una “caja negra”. Esto significa que, aunque hagan bien su trabajo, normalmente no sabemos muy bien por qué el modelo toma determinadas decisiones.
La buena noticia es que se han desarrollado recientemente nuevos métodos capaces de abrir esta “caja negra”. Existen técnicas de interpretación como Shapley y LIME que pueden mostrar el impacto que tienen determinadas entradas. Cuando las huellas moleculares utilizadas para entrenar el modelo están basadas en subestructuras, como las claves MACCS o las huellas de subestructuras Pubchem, estas técnicas de interpretación pueden enseñarnos directamente el efecto que la presencia o ausencia de determinadas subestructuras en las moléculas puede tener sobre su actividad predicha. Este conocimiento es muy poderoso, porque puede ayudarnos a guiar el desarrollo de compuestos hacia inhibidores más potentes.
Para facilitar y agilizar todo este proceso, nuestro grupo de investigación ha desarrollado Sibila, una herramienta de línea de comandos que permite entrenar distintos modelos de aprendizaje automático y aplicar sobre ellos diferentes técnicas de interpretación de manera automática. Para aumentar la fiabilidad de las técnicas de interpretación, se entrenan diferentes modelos de aprendizaje automático sobre los datos. Una vez entrenados estos modelos, aplicamos técnicas de interpretación como LIME y Shapley. ¿Y cómo sabemos cuáles características de entrada son las más importantes? Mediante el análisis de los resultados de interpretación de todos los modelos. Una vez identificadas las características más relevantes mediante este método de consenso, los resultados pueden visualizarse fácilmente con un script que he escrito. Esto puede ayudarnos a ver con más claridad las subestructuras y cómo pueden influir en la bioactividad del compuesto.
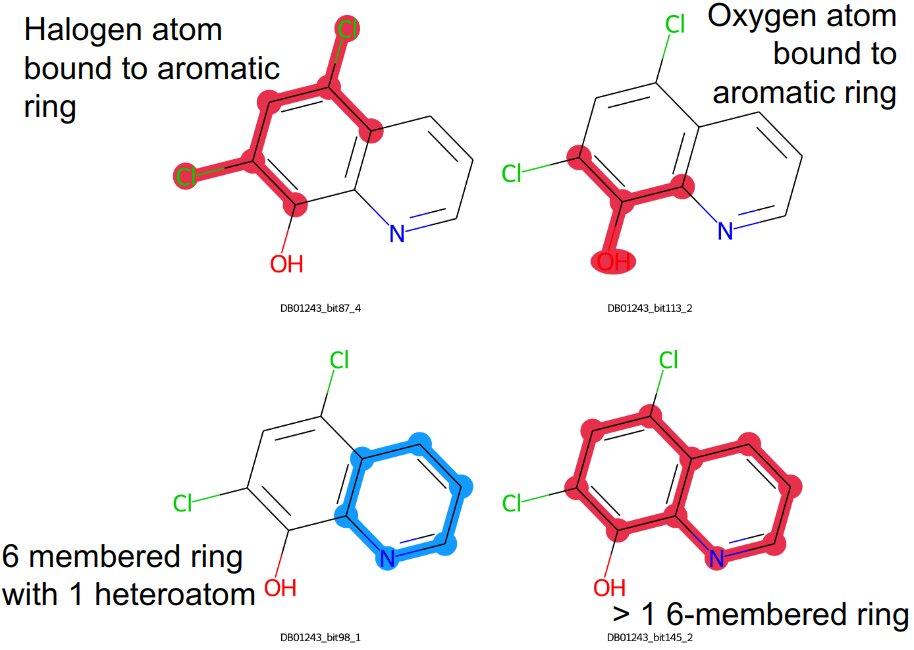
Un ejemplo de subestructuras relevantes visualizadas junto con su efecto. Cuando la subestructura está resaltada en rojo, tiene un efecto positivo en la actividad prevista, mientras que el color azul significa lo contrario. Utilizando esta información, se puede intentar incluir más subestructuras buenas, al tiempo que se intenta eliminar las que tienen un efecto negativo. En este caso, el anillo con un heteroátomo tiene un efecto negativo, por lo que podría mejorarse cambiando el nitrógeno por un carbono o añadiendo otros heteroátomos para que no haya solamente un heteroátomo.
En la imagen 2 se muestra un ejemplo. Como se puede ver, tenemos una estructura química, y sobre ella se han visualizado 4 características de la huella MACCS. Si la subestructura está resaltada en rojo, significa que tiene un efecto positivo en la actividad, mientras que si la subestructura está resaltada en azul, tiene un impacto negativo en la actividad. En este caso, la molécula tiene 3 subestructuras que tienen un impacto positivo en la actividad, pero una de ellas tiene un impacto negativo. En este caso, esto se debe a que la molécula tiene un anillo con 1 heteroátomo. Esto sugiere que si otros átomos de este anillo también fueran heteroátomos, o si el nitrógeno fuera reemplazado por un átomo de carbono convencional, la actividad de este compuesto podría potencialmente mejorar. De este modo, podemos utilizar estas interpretaciones para guiar el diseño de nuevos compuestos con el fin de mejorar la actividad.
Como puede verse, el futuro del descubrimiento de fármacos mediante el cribado virtual basado en el aprendizaje automático es prometedor. Mediante el uso de técnicas de huella molecular e interpretación, podemos entender y visualizar cómo determinadas subestructuras pueden influir en la actividad inhibidora del compuesto. Esto puede ayudarnos a desarrollar inhibidores más potentes y específicos. Además, con diversas técnicas de interpretación y aprendizaje automático, podemos confiar más en nuestros hallazgos. Con el desarrollo de herramientas como Sibila, ahora cualquier persona puede entrenar e interpretar estos modelos. En nuestros proyectos anteriores, hemos demostrado que estas herramientas pueden ayudarnos a descubrir nuevas clases de compuestos en diferentes contextos de descubrimiento de fármacos. De esta forma, esperamos que estos avances sirvan de inspiración a otros investigadores de las ciencias químicas para descubrir nuevas químicas y desarrollar fármacos más potentes.