Hace mucho tiempo, unos 25 años, en un lugar muy lejano, véase Alemania, a unos investigadores húngaros, Zsuzsanna Izsvák y Zoltán Ivics, se les ocurrió investigar en el semen del salmón un antiguo gen dormido, inactivado por muchos milenios, con el objetivo de conseguir salmones enormes que sirvieran para la pesca deportiva. Tal vez no fuera una meta especialmente ambiciosa, pero en cuanto consiguieron despertarlo de su sueño, los científicos no pararon de encontrarle utilidades. Se trataba de un transposón (o gen saltarín, para los amigos), un tipo de elemento genético que no hacía apenas cuarenta años había sido descubierto por la citogenetista Bárbara McClintock mientras estudiaba el color de los granos del maíz. Y tuvieron Izsvák e Ivics el acierto de llamarlo Sleeping Beauty, o menos cariñosamente transposón SB. Pero, ¿qué hacía dormido el gen de la Bella Durmiente? ¿Para qué sirve en realidad? ¿Qué tantas utilidades puede tener y qué tiene que ver todo esto del semen de salmón con el cáncer? ¿De verdad está permitido poner nombres tan frikis a las moléculas?
Primero, una breve historia de los frikismos en Genética
 La Bella Durmiente se llama así por ser el primer transposón que, tras millones de años dormido en un genoma, fue “despertado” tras su siesta. En Genética es bastante común encontrarse, en mitad de mamotretos infumables y artículos tremendamente áridos de leer, nombres que parece que el que bautizó a su descubrimiento estaba tratando de tomarnos el pelo. Y es que los científicos nos aburrimos mucho y los noventa, época de grandes descubrimientos en Genética, hicieron mucho daño. Si es necesario dar ejemplos, podemos encontrarnos genes con nombres de personajes de videojuegos, como el gen sonic hedgehog (erizo Sonic), que se encuentra en el genoma de las moscas de la fruta (Drosophila melanogaster) y cuya mutación produce embriones con pelos de aspecto de erizo.
La Bella Durmiente se llama así por ser el primer transposón que, tras millones de años dormido en un genoma, fue “despertado” tras su siesta. En Genética es bastante común encontrarse, en mitad de mamotretos infumables y artículos tremendamente áridos de leer, nombres que parece que el que bautizó a su descubrimiento estaba tratando de tomarnos el pelo. Y es que los científicos nos aburrimos mucho y los noventa, época de grandes descubrimientos en Genética, hicieron mucho daño. Si es necesario dar ejemplos, podemos encontrarnos genes con nombres de personajes de videojuegos, como el gen sonic hedgehog (erizo Sonic), que se encuentra en el genoma de las moscas de la fruta (Drosophila melanogaster) y cuya mutación produce embriones con pelos de aspecto de erizo.
También tenemos el gen de la pikachurina, una proteína relacionada con la transmisión de impulsos eléctricos en las neuronas, o el oncogén Zbtb7, inicialmente conocido como gen Pokemon y relacionado, como buen oncogén, con la activación de fenómenos cancerosos cuando se estropea. A su descubridor principal, el genetista italiano Pier Paolo Pandolfi, se le ocurrió la brillante idea de llamarlo así como abreviación de “POK erythroid myeloid ontogenic factor” o “factor eritroide mieloide ontogénico de la proteína POK”, pero no le duró mucho la broma porque enseguida la compañía de PokemonTM puso una demanda ante los titulares de “Pokemon da cáncer” y se le cambió el nombre por uno un poco más prosaico en relación a su trabajo como factor con región “dedo de zinc” (una estructura típica de proteínas que tienen funciones muy importantes, claves, regulando la expresión de los genes).
Otros genes tienen nombres que juegan con su función para con otros genes. El gen de la proteína Smaug, por ejemplo, recibe su nombre del dragón inventado por Tolkien porque mantiene a raya, durante el desarrollo embrionario de las moscas de la fruta, a los productos génicos del gen nanos (en alusión a que en la historia de Tolkien, los enanos intentaban recuperar su fortaleza de las garras del dragón Smaug). La mutación en el gen áspid, por su parte, también en la mosca de la fruta, es tremendamente letal, pero sólo si coincide en el genoma con la mutación del gen cleopatra. Lo sabemos: sólo los genetistas podrían tener una mente así. Y algunos se lo curran directamente nada, como cuando se bautizó a un gen (que, más tarde, daría nombre a una familia entera de genes) como RING. Este gen, como sus parientes (que se pueden contar por cientos en el ser humano), también da proteínas con dedos de zinc, pero su nombre no se refiere a nada de eso: sólo son las siglas de “Really Interesting New Gene” (gen nuevo realmente interesante). Todo un portento de la creatividad.
Dentro de esta familia de genes RING, tenemos al gen Ariadne-1 (ari-1), descubierto en los 2000 y postulado como un gen crucial para el desarrollo embrionario del sistema nervioso y muscular y la diferenciación celular en general en organismos desde las moscas hasta los seres humanos, de la misma manera que Ariadna fue la que ayudó a Teseo con su hilo mágico guía a salir del laberinto del minotauro. En fin, la lista es infinita. En el fondo, a todos nos gusta pasarlo bien en el trabajo, ¿no?
Segundo, una review de los genes saltarines
Los genes saltarines son de esos descubrimientos que lo cambian todo, aunque no son muy conocidos fuera del ámbito científico. En 1983, supusieron el premio Nobel para la investigadora estadounidense Barbara McClintock, tras muchos años de ignorar y dar la espalda a su trabajo. Resumidamente, su hallazgo radicó en buscar la explicación a por qué en algunos granos de maíz la corteza (capa de aleurona) tenía manchas de color oscuro. McClintock, determinó por puro análisis genético clásico, sin utilizar herramientas moleculares como la PCR ni nada por el estilo, la existencia de dos genes (en su terminología, mejor, “elementos reguladores”), que vino a llamar activador (Ac) y disociador (Ds). Estudió varias generaciones de mazorcas de maíz y llegó a la conclusión de que tanto Ac como Ds son elementos transponibles, es decir, genes o trozos de ADN que pueden cambiar su posición en el genoma, pasando de unos cromosomas a otros. Si el gen Ds está colocado en medio del gen que determina pigmentación en la aleurona del maíz, entonces el gen pigmentador está apagado, interrumpido, no funciona, de manera que la capa se ve transparente y tenemos un grano amarillo. Sin embargo, cuando el gen Ac está presente, su proteína permite al gen Ds despegarse de su sitio y colocarse en otro lugar, dejando el gen pigmentario constituido de nuevo.
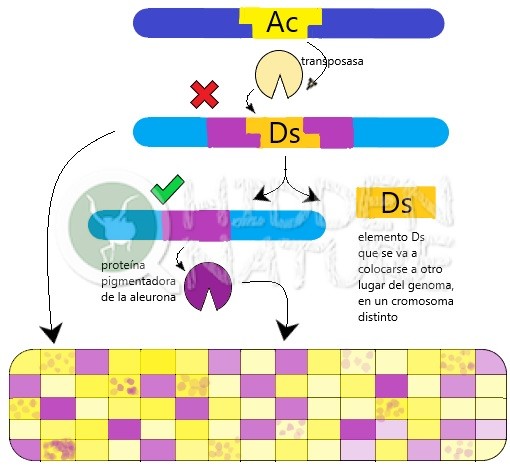
En este esquema pretendemos mostrarte cómo el elemento Ac, que codifica la transposasa, puede movilizar al gen saltatín Ds cuando está presente. En tal caso, el gen Ds se desprende del gen pigmentador al que interrumpe, dando pie a que las células presenten pigmento y el grano de maíz esté coloreado. En caso contrario, no se produce ese pigmento y resultará un grano amarillo, de capa de aleurona transparente. Algunos tienen manchas pigmentadas porque Ds se desprende del gen durante el desarrollo del grano en algunas células, dando lugar a poblaciones celulares con el gen pigmentador activado, mientras que otras lo mantienen interrumpido
Cabe anotar que hay dos tipos de transposones, los de ARN, llamados “de copia y pega”, y los de ADN, o “de corta y pega”. Estos últimos, que son los que nos atañen, constan de una secuencia repetida en los extremos, y en medio una secuencia que codifica para una proteína, la enzima transposasa. Una vez expresada, esta enzima se une a las secuencias repetidas de ADN del extremo del transposón y lo escinde del resto del genoma, reintegrándolo posteriormente en otra zona, no importa mucho dónde. Así, volviendo a McClintock, mientras que Ac puede moverse de forma independiente (es un elemento autónomo), Ds puede moverse a otro lugar del cromosoma sólo bajo la influencia de Ac, que es el elemento que puede sintetizar transposasa. Durante el desarrollo de un grano de maíz que contenga ambos elementos transponibles, puede suceder que, en algunas células, de forma arbitraria, el gen Ds se descoloque de su sitio y deje de interrumpir al gen pigmentador, con lo que las células hijas de ésta puedan expresar pigmento violeta. Tendremos, pues, un grano amarillo a parches violeta, que serán tanto más grandes cuanto más temprano haya ocurrido la escisión del gen Ds durante el desarrollo del grano.
Puede parecer una anécdota curiosa sin más, pero prácticamente todos los organismos investigados, en realidad, tienen secuencias que pueden moverse de un lado a otro de su genoma y que pueden llegar a representar un porcentaje muy alto del mismo: en Drosophila sólo constituyen un 10%, pero hasta un 45% del genoma del ser humano está formado por elementos transponibles, y el del maíz, hasta un 50%. La mayoría de los elementos transponibles pueden insertarse en muchos sitios diferentes sin necesidad de que exista homología entre sus secuencias y con frecuencia estos elementos producen mutaciones, ya sea porque se insertan en otro gen, porque lo rompen al escindirse de él o porque inducen reordenamientos en la secuencia de ADN (deleciones, duplicaciones, inversiones, etc.).
¿Para qué existen estos transposones? Es decir, ¿por qué tenemos estos “genes saltarines” cuya única función es producir una proteína que los cambia de sitio? Pues, en realidad, para nada. No son nuestros. Se interpreta que son parásitos moleculares que van por su cuenta, como si en una biblioteca ordenadita se hubieran colado libros que no sólo no pertenecen a la biblioteca, sino que van cambiando de sitio aleatoriamente. Hay estimaciones que dicen que tenemos diez veces más elementos transponibles en nuestro genoma que genes humanos con función propia. Pero no te preocupes: están prácticamente todos dormidos. La evolución los ha inactivado todos porque, de lo contrario causarían demasiados problemas. la mayoría de ellos están inactivados por mutaciones que han alterado sus transposasas o las secuencias en las que esta corta para despegar el transposón. Podemos usarlos, de hecho, para rastrear la evolución de las especies, estudiando los transposones “fósiles”, inutilizados en el genoma de las especies emparentadas para ver cuáles tenemos comunes y cuáles no y seguir, así, la pista de nuestro parentesco.
Tercero, sobre la Bella Durmiente combatiendo los tumores

Una vez generado un tumor, sólo hay que buscar los transposones y ver en qué genes se han insertado para encontrar candidatos. Como los ratones y los humanos somos genéticamente muy similares por el parentesco que nos une, muchos de estos genes son parecidos a los humanos, tal que encontrando en el ratón qué secuencias de ADN actúan como pro-cáncer cuando se interrumpen, podemos empezar a estudiarlos en los humanos y ver si podemos suplir la carencia de este gen de forma artificial. Es muy importante saber qué genes son los que conducen a la formación de los tumores para poder diseñar mejor las estrategias terapéuticas.
Se han diseñado transposasas SB mejoradas, más eficaces, que pueden responder incluso a sustancias que nosotros añadamos, y se ha usado este elemento como vector para introducir genes en genomas que queremos estudiar. Se trata de un sistema prometedor para la biomedicina y para la investigación genética del futuro, insertando genes terapéuticos en humanos con enfermedades genéticas, permitiendo estudiar mejor rutas de expresión génica y señalización celular, generar mejores transgénicos, etc. Parece mentira que alguien pudiera estar muy interesado en saber cómo los granos de maíz determinan su color, pero es tanto más asombroso que de ahí haya salido semejante campo de estudio. Tampoco nadie se esperaría que despertar un trozo de ADN parásito dormido dentro del genoma del salmón, a partir de unas muestras de semen, tuviera alguna relación con la investigación oncológica y la biotecnología… Pero, a fin de cuentas, así es como funciona la tan denostada investigación en ciencia básica: no es necesario que algo tenga una aplicación obvia a un proceso mercantilizable ni necesariamente un hallazgo queda restringido a la idea original que motivaba su búsqueda. Nunca se sabe. Estas historia, cuantas veces las contemos, tienen siempre la misma moraleja.
FUENTES Y REFERENCIAS
- BROWN, T.A. (2008) Genomas (3ª ed.). Médica Panamericana, Buenos Aires
- HENDERSON, M. (2010) 50 cosas que hay que saber sobre Genética. Ariel
- IVICS, Z.; IZSVÁK, Z. (2010) The expanding universe of transposon technologies for gene and cell engineering. Mobile DNA, 1(1):25 | DOI: 10.1186/1759-8753-1-25
- AGUILERA, M.; OLIVEROS, M.; MARTÍNEZ-PADRÓN, M.; BARBAS, J.A.; FERRÚS, A. (2000) Ariadne-1: a vital Drosophila gene is required in development and defines a new conserved family of ring-finger proteins. Genetics, 155(33). pp: 1231-44 | PMID: 10880484, PMCID: PMC1461160
- BORDEN K.L.; FREEMONT, P.S. (1996) The RING finger domain: a recent example of a sequence-structure family. Current Opinion in Structural Biology, 6 (3). pp: 395–401 | doi:10.1016/S0959-440X(96)80060-1
- COLLIER, L.S.; CARLSON, C.M.; RAVIMOHAN, S.; DUPUY, A.J.; LARGAESPADA, D.A. (2005) Cancer gene discovery in solid tumours using transposon-based somatic mutagenesis in the mouse. Nature, 436. pp:272–6.

