Las enfermedades raras, por definición, son aquellas cuya prevalencia es inferior a 5 casos por cada 10.000 personas. Lo que quiere decir que son enfermedades menos frecuentes que aquellas consideradas dentro la norma, término que estadísticamente responde simplemente a aquello que se da en mayor proporción.
En los últimos años, muchas de estas enfermedades o síndromes son conocidas por la sociedad gracias a asociaciones creadas por afectados y familiares, que se movilizan para hacer saber que existen necesidades derivadas de las mismas que no están cubiertas. Podemos así nombrar, sin que nos resulten totalmente ajenas, el autismo hereditario, el Síndrome de Williams, el de Prader-Willis, el de Marfan, la Esclerosis Lateral Amiotrófica (ELA), y un larguísimo etcétera. La gran mayoría de enfermedades raras aparecen antes de los 2 años y en un alto porcentaje tienen una base genética. Esto, en la cotidianidad, se traduce en pacientes de muy temprana edad, con dolencias sin un diagnóstico sencillo ni inmediato.
Como todos los seres vivos, las características que presentamos vienen en primaria instancia marcadas por la información que está escrita en nuestros genes, y no es una forma de hablar. Nuestra propia esencia la leemos con letras (nucleótidos nombrados por sus base nitrogenada, es decir, Adenina [A], Citosina [C], Guanina [G] o Timina [T]). Es nuestro genotipo, utilizando el término en su sentido más general.
Esta información, que guarda nuestro ADN, no es idéntica entre personas y, en ocasiones, puede presentar pequeños “errores” en uno o múltiples genes que deriven en el desarrollo de una enfermedad rara. Esto hace que el diagnóstico de las mismas no se pueda realizar con las pruebas más habituales en el sistema sanitario. Aquí entramos en el mundo del diagnóstico genético.
Como en cualquier otro diagnóstico médico, lo que esperamos obtener cuando somos los afectados es una respuesta sobre qué origina nuestra enfermedad. Pero en los pacientes, la información obtenida no se limita solo a ellos, si no que se amplía a familiares biológicos. Así podemos deducir si se trata de una enfermedad de carácter hereditario o bien es de novo, si es una herencia de origen paterno, materno o ambos, qué probabilidades hay de tener más hijos con la misma dolencia, u obtener esta información a edades muy tempranas, incluso, prenatales. En los protocolos de diagnóstico genético se va avanzando casi de la mano de la investigación básica, por lo que si bien es todavía relativamente bajo el nivel de estandarización, tienen un alto dinamismo en sus actualizaciones.
Desde la década de los 70 del pasado siglo, las técnicas para poder secuenciar ADN, es decir, la manera de poder leer nuestra información genética, han avanzado a una velocidad y con un alcance asombroso, hasta pasar de los métodos más básicos a las actualmente conocidas como NGS (Next Generation Sequencing). De una manera coloquial, podríamos decir que hemos pasado de leer una página de un libro para niños de 3 años en meses, a descifrar el Quijote en horas.
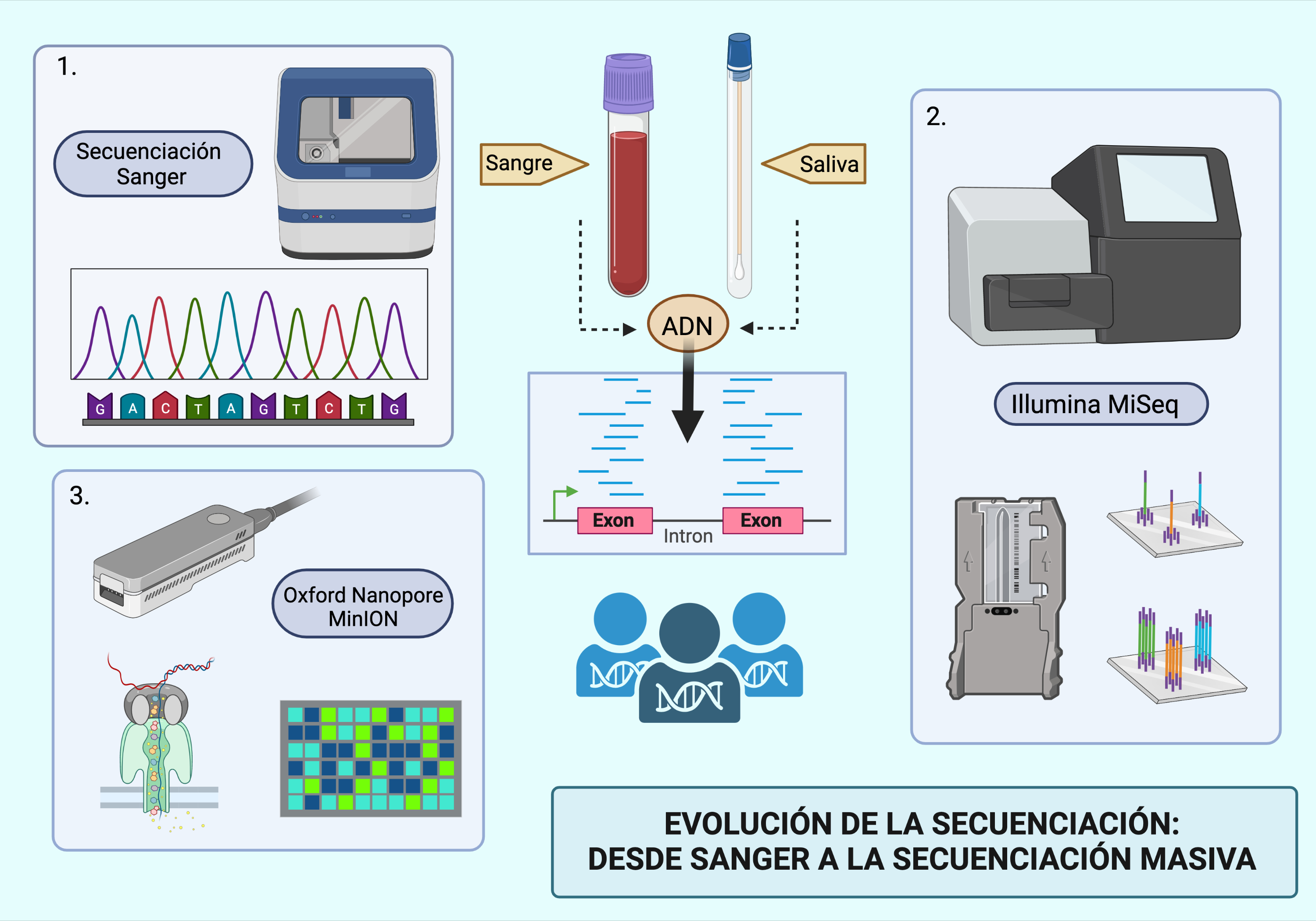
Esquema realizado con la plataforma Bio Render de tres diferentes tecnologías de secuenciación. (1) Método Sanger para la secuenciación de ADN. Fue una de las primeras tecnologías desarrolladas pero todavía se utiliza hoy en día. Desarrollado por el bioquímico Frederick Sanger (premio Nobel de Química en 1980). La comercialización del primer secuenciador automático del método Sanger fue en 1986. Permitía obtener unas 200 bases cada hora, de una sola muestra, visualizadas en forma de electroferograma. Este tipo de secuenciadores de primera generación fueron empleados para secuenciar el primer genoma humano. (2 ) Ya en la época de surgimiento de la llamada Next Generation Sequencing (NGS) se desarrollaron los secuenciadores llamados de segunda generación, entre ellos los de la plataforma Illumina. En 2006 salió a la venta el primero de este tipo. Tienen la capacidad de realizar múltiples secuencias en paralelo, generando millones de lecturas al mismo tiempo; Su mejora principal es usar una superficie sólida dese fijación, esto permite que se utilice mucha menor cantidad de reactivos y se abarata el coste por nucleótido. (3) Secuenciadores de tercera generación como la tecnología desarrollada por Oxford Nanopore. Con un secuenciador que cabe en la palma de una mano se pueden secuenciar largas cadenas de material genético. Se lleva a cabo la secuenciación en tiempo real mediante la detección de un cambio de voltaje producido por el paso de la secuencia de ADN por un poro.
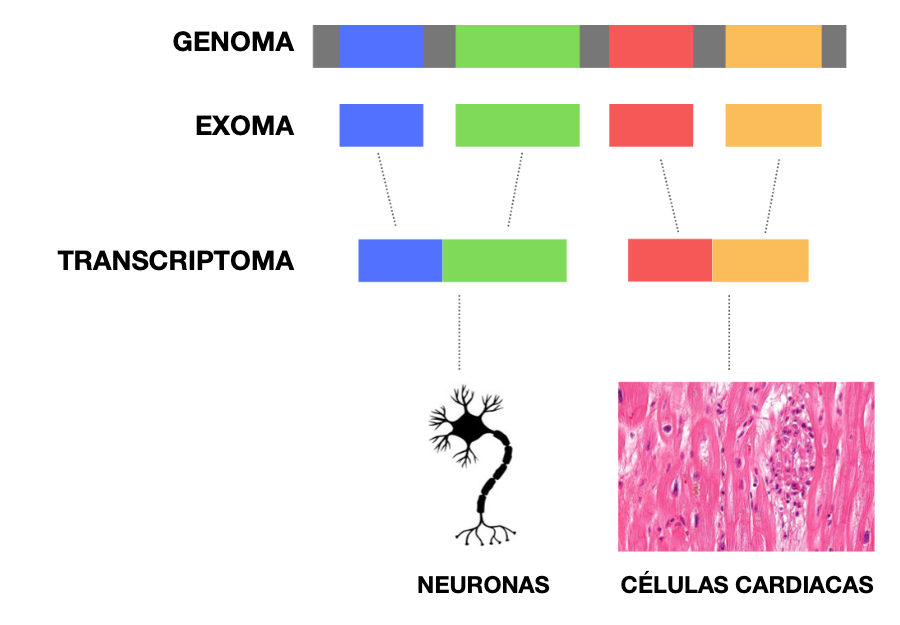
Con todos estos avances a nuestro alcance, actualmente en el laboratorio se realiza lo que se conoce como una secuenciación del exoma. Secuenciar un exoma significa centrarse en obtener sólo las secuencias de aquellas partes de todo nuestro genoma que son codificantes, exones, y dejar de lado aquellas que no, denominadas intrones. Ser codificante implica que esa región del genoma se va a transcribir y traducir dando lugar a proteínas funcionales. Los exones ocupan menos del 2% de todo el genoma. Y…¿qué ventajas tiene esta opción? De entrada podríamos destacar que se trata de una prueba muy homogénea, es decir, partiendo de un poco de sangre, o incluso saliva, se sigue el mismo procedimiento para cualquier paciente que necesite diagnóstico genético. Es un punto destacable, puesto que la mayoría de las enfermedades raras no tienen un cuadro sintomatológico cerrado, ni todos los pacientes tienen un mismo historial clínico. Ni siquiera todos los afectados por una misma enfermedad presentan un mismo fenotipo (rasgos o características físicas observables). Además, obtener el exoma completo nos permite no analizar simplemente aquellos genes que sabemos que están relacionados con determinadas dolencias, sino identificar variaciones que aún no han sido descritas ni ligadas a ninguna enfermedad rara. Pero las ventajas no terminan aquí: nuestro exoma es un libro ya escrito que se puede volver a leer y revisar tantas veces como necesitemos, a medida que la ciencia vaya incorporando conocimiento en este ámbito. Será como tener una base de datos personal e intransferible en la que podamos buscar nuevas variaciones siempre que nuestros médicos lo consideren oportuno.
Como ocurre muchas veces en temas de salud, es triste pero inevitable ligar las posibilidades de realizar pruebas a un coste, tanto de tiempo como de recursos. Y aquí avanzamos en la buena dirección; cuando se terminó de secuenciar el primer genoma humano, se habían necesitado 10 años y se habían invertido más de 2000 millones de dólares. Hoy en día, con un dispositivo que cabe en la palma de una mano, podemos obtener un exoma completo en 30 minutos. Toda esta tecnología de secuenciación necesita de un soporte bioinformático, ya que es tan esencial obtener información como poder procesarla e interpretarla. En cierta manera, estas áreas de la ciencia que hace pocos años parecían propias de una película de ciencia-ficción, se han incorporado a la rutina de los laboratorios de investigación que se dedican a estos campos. La transferencia de la metodología al campo clínico es muy inmediata, y su optimización ha permitido reducir costes del orden de 1000 veces por base secuenciada, y el nacimiento de empresas y tecnologías dirigidas exclusivamente a ello.
Llegados a este punto debemos ser conscientes de que disponemos de recursos humanos con la formación necesaria para realizar diagnóstico y consejo genético, así como tecnología a nuestra disposición para obtener toda la información que necesitamos. He aquí el papel clave que tenemos que asumir como sociedad. Podemos permitirnos, y debemos exigirnos, incorporar al circuito de salud estos servicios de diagnóstico, como otros ya existentes. Los afectados por enfermedades raras deberían seguir el mismo itinerario que cualquier otro paciente, sin tener que padecer más por un simple hecho estadístico.